其实有些时候,我们将SEM想的过于复杂了,其实操作起来还是较容易上手的,不过建模过程中需要我们根据自己试验设计等自行建立一个因子间的关系模型,然后对这个模型进行反复调试,直至达到自己满意的结果为止,这就是SEM的难点,因为构建这个关系模型需要丰富的经验,但是有没有什么入门比较快的法门呢,当然是有的——借鉴前人的文献!!!一般建模前,我们会通过一些相关性分析、VIF、CCA/RDA等筛选一下用于建模的因子,去除不必要的因子,使得起始模型的建立更简单一些,也可以通过相关性结合研究实际初步评估一下直接作用和间接作用。
看看别人家的SEM
写论文的过程中参考文献的查阅是必不可少的,我们会根据自己的研究方向找到与自己试验设计比较契合的文献,看看他们的SEM是这么做的,那我们照着前辈们文献中的SEM模型照猫画虎,一般拟合结果都会八九不离十。小编是做草地生态的,就查一些和草地生态有关的SEM看看吧。
我们就以最后一篇文章的结构方程模型为例来照猫画虎,首先来研究一下这个结构方程模型的组成成分,这个结构方程模型中地上生物量、土壤物理性质、土壤化学性质之间是双向箭头,也就是说它们之间是相互影响的,然后地上生物量、土壤物理性质、土壤化学性质又分别指向不同的指标,表明这三个因素又分别受这些指标的影响。接下来让我们看看怎么在R中表示它们之间不同的关系。
基本语法简介
语法一:f3~f1+f2(路径模型)
结构方程模型的路径部分可以看作是一个回归方程。而在R中,回归方程可以表示为y~ax1+bx2+c,“~”的左边的是因变量,右边是自变量,“+”把多个自变量组合在一起。那么把y看作是内生潜变量,把x看作是外生潜变量,略去截距,就构成了结构方程模型的语法一。
语法二:f1 =~ item1 + item2 + item3(测量模型)
“=~”的左边是潜变量,右边是观测变量,整句理解为潜变量f1由观测变量item1、item2和item3表现。
语法三:item1 ~~ item1 , item1 ~~ item2
“~~”的两边相同,表示该变量的方差,不同的话表示两者的协方差
语法四:f1 ~ 1
表示截距
此外还有其它高阶的语法,详见lavaan的help文档,一般的结构方程建模分析用不到,便不再列出。
SEM实现代码
采用完全描述的方法指定所有要素
需要调用的R包:lavaan (结构方程建模),semPlot (结果可视化)
示例数据排列方式如下:
关注公众号“红皇后学术”回复“SEM”即可下载完整代码与示例数据!!!
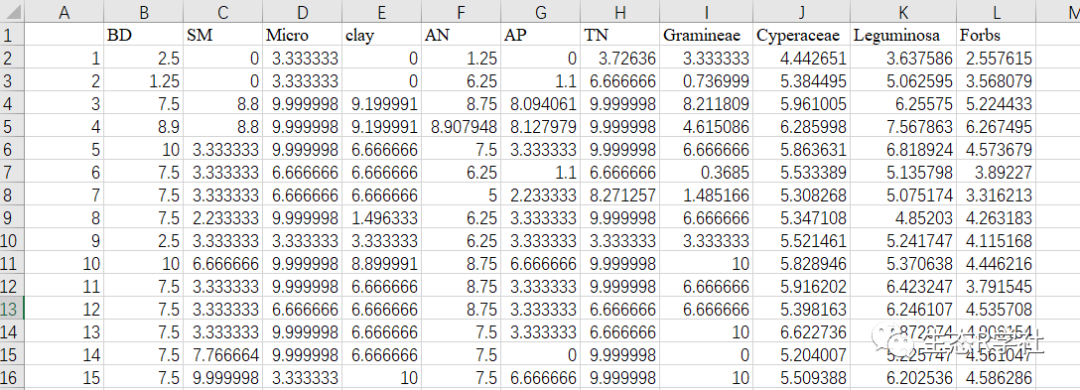
构建模型框架
#调用用于建模的R包library(lavaan)#读取自己的数据Mydata<-read.csv(file.choose(),header=T, row.names = 1)head(Mydata)#第一步,照猫画虎构建文章4中所示的模型model<- '# 利用被测变量(右)定义潜在变量(左):测量模型。意思就是这几个指标构成了它们Above ground biomass =~ Gramineae + Cyperaceae + Leguminosa + ForbsPhysical properties =~ BD + SM + Micro + clayChemical properties =~AN + AP +TN# 构建回归方程:路径模型。各测量模型之间的关系,本文中它们是两两相互影响的Above ground biomass ~~ Physical propertiesAbove ground biomass ~~ Chemical propertiesPhysical properties ~~ Chemical properties# 指标间的关系,两边相同,表示该变量的方差,不同的话表示两者的协方差,文章4中并未描述指标间的方差或者协方差,模仿出文章4的结构方程模型上两步就够了,如果想做进一步分析可进行这一步,为了更好的讲解,本文就顺便描述一下变量间的方差Gramineae ~~ CyperaceaeLeguminosa ~~ ForbsBD ~~ SMAN ~~ AP
拟合模型
#适配模型fit <- sem(model, data = Mydata)#查看拟合结果summary(fit)summary(fit, fit.measures=TRUE)summary(fit, standardized = TRUE)

潜在变量的路径系数大小
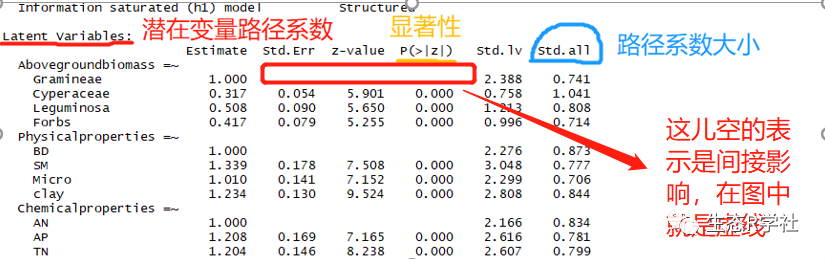
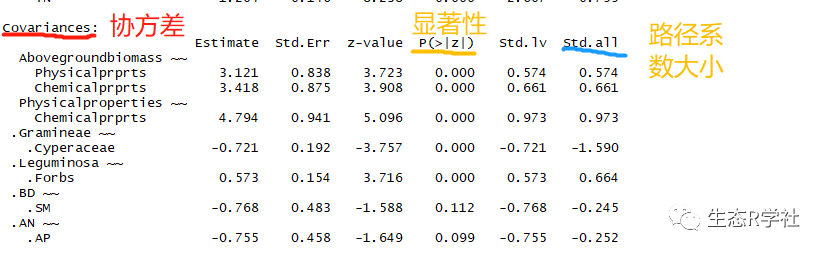
协方差的路径系数大小
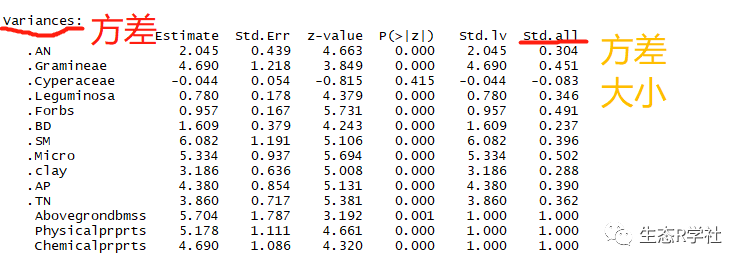
方差的路径系数大小
计算拟合系数
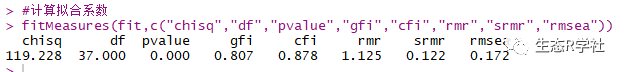
#计算拟合系数fitMeasures(fit,c("chisq","df","pvalue","gfi","cfi","rmr","srmr","rmsea"))
拟合系数结果如下:
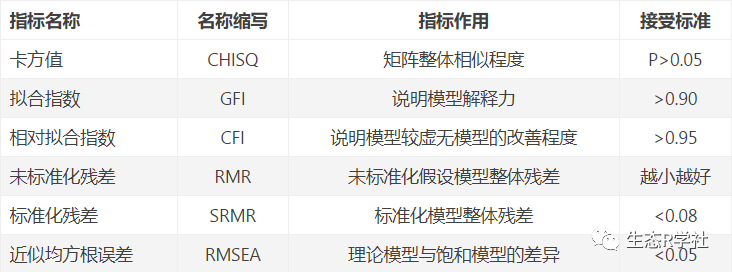
拟合系数结果的接受标准
结果的可视化
#调用SEM绘图包library(semPlot)semPaths(fit)semPaths(fit, what = "std", layout = "tree2", fade=F, nCharNodes = 0)semPaths(fit, what = "std", layout = "tree2", fade=F, nCharNodes = 0, intercepts = F, residuals = F, thresholds = F)
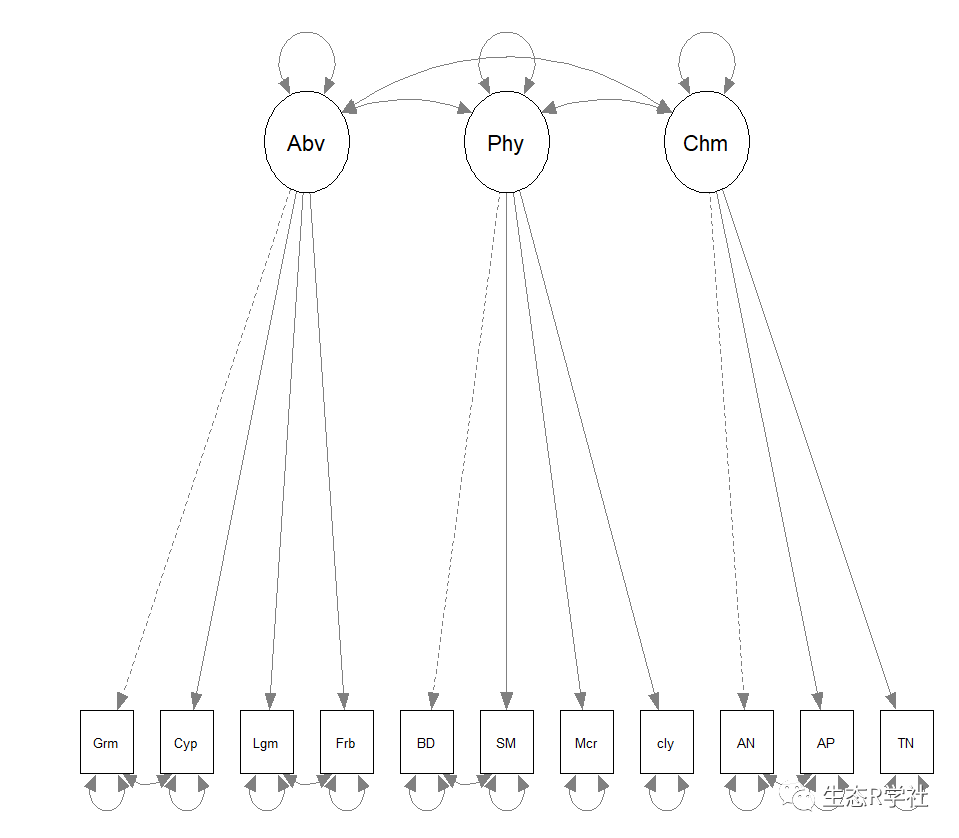
semPaths(fit, what = "std", layout = "tree2", fade=F, nCharNodes = 0)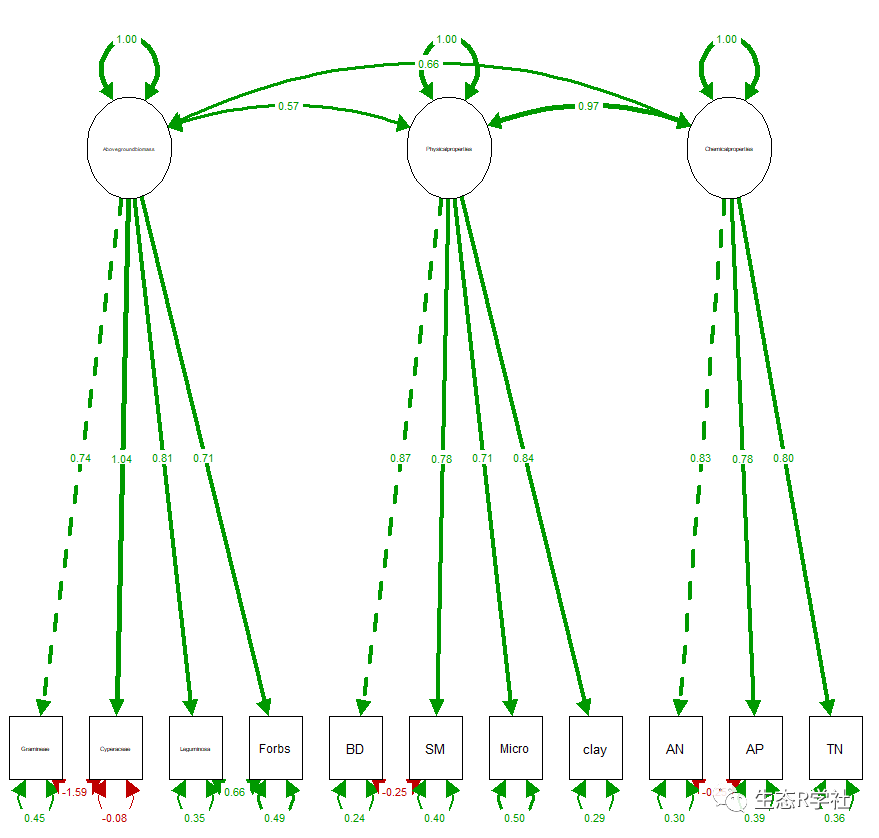
semPaths(fit, what = "std", layout = "tree2", fade=F, nCharNodes = 0, intercepts = F, residuals = F, thresholds = F)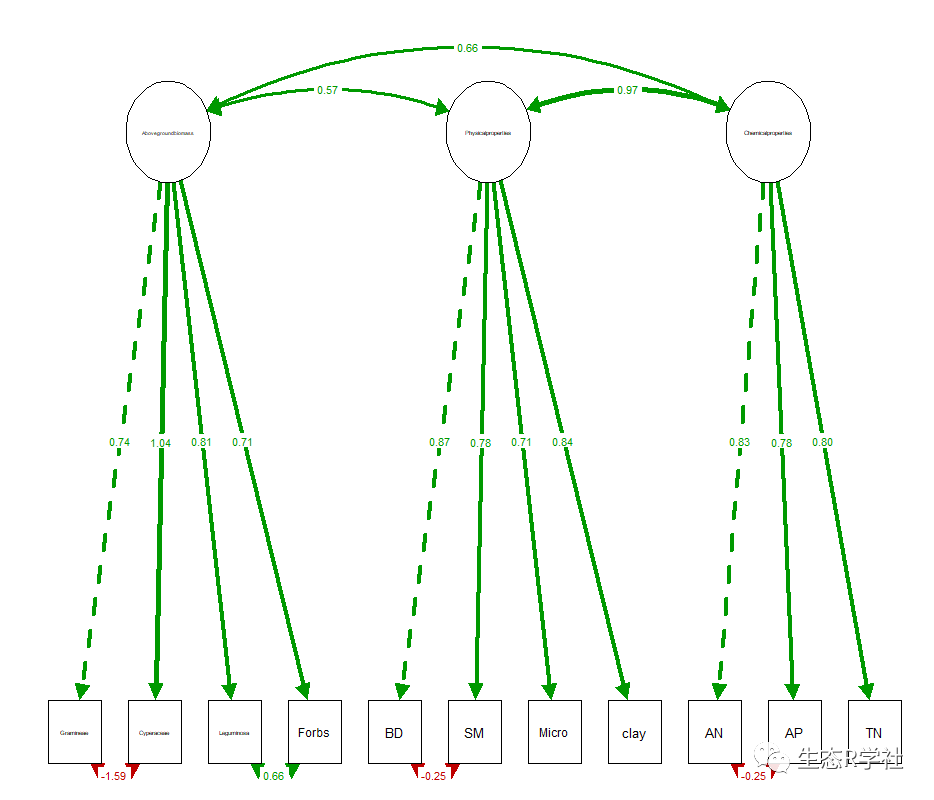
semPaths(fit,layout = "circle", fade=F, nCharNodes = 0)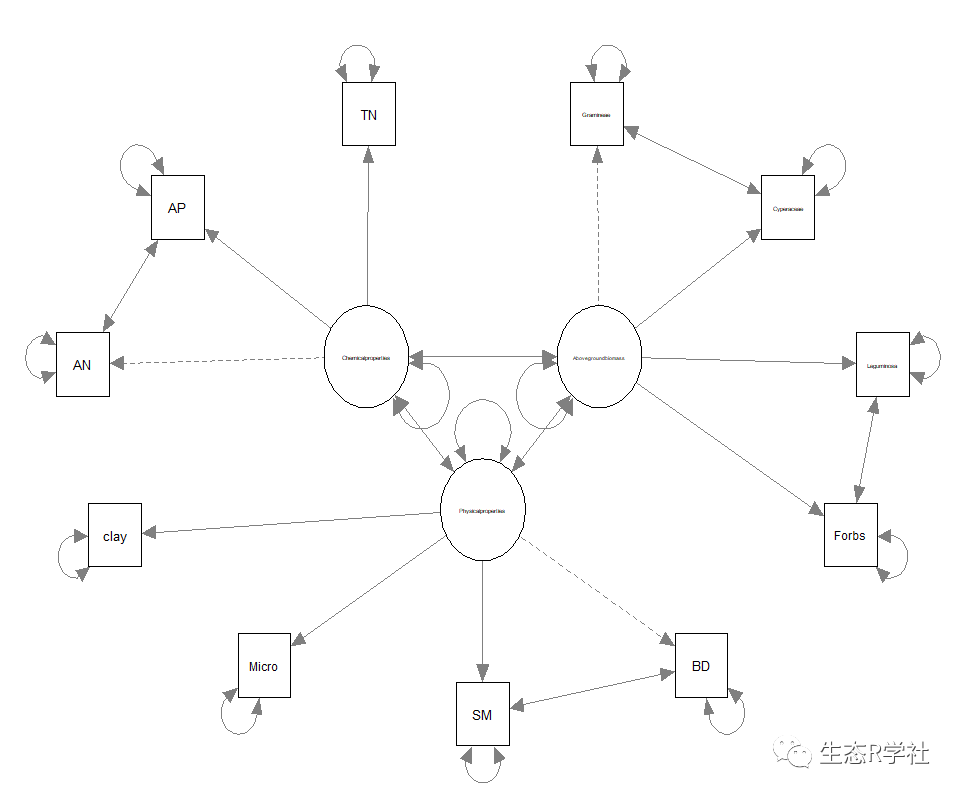
semPaths(fit,layout = "spring")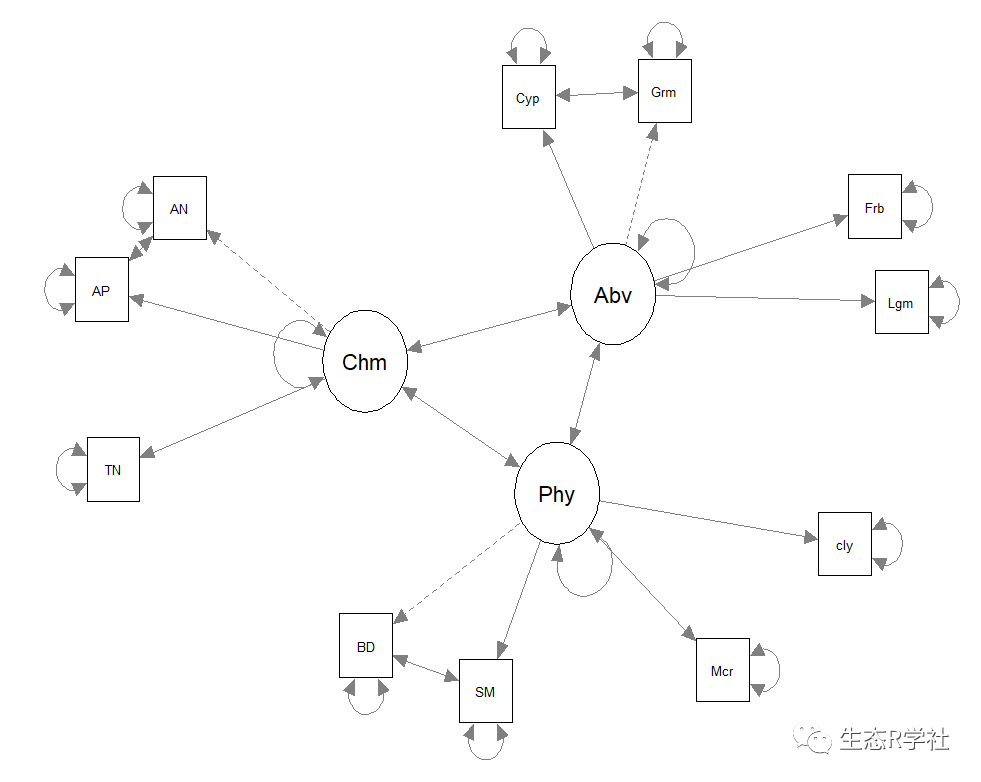
写在最后
不得不说,做出来的图确实有些难看,但是一般情况下只要求得模型路径系数大小以及直接、间接的影响关系即可,剩余的我们便可以发挥自己的想象力去自己美化最后得出的结构方程模型。